-
Notifications
You must be signed in to change notification settings - Fork 4
Cómo evaluar calibre de axones
Cómo evaluar el calibre de axones
Esto se logra a partir de histología. En este ejemplo, trabajaremos con una imagen con tinción de azul de toluidina, con un grosor de 1 micra, y con un aumento de 100X. Esta imagen es cortesía del Dr. Larriva, y se trata de un nervio óptico de rata, cortado de manera perpendicular al eje longitudinal de los axones.
La imagen original está en tif. La que se anexa aquí está en png por requisitos del dokuwiki.
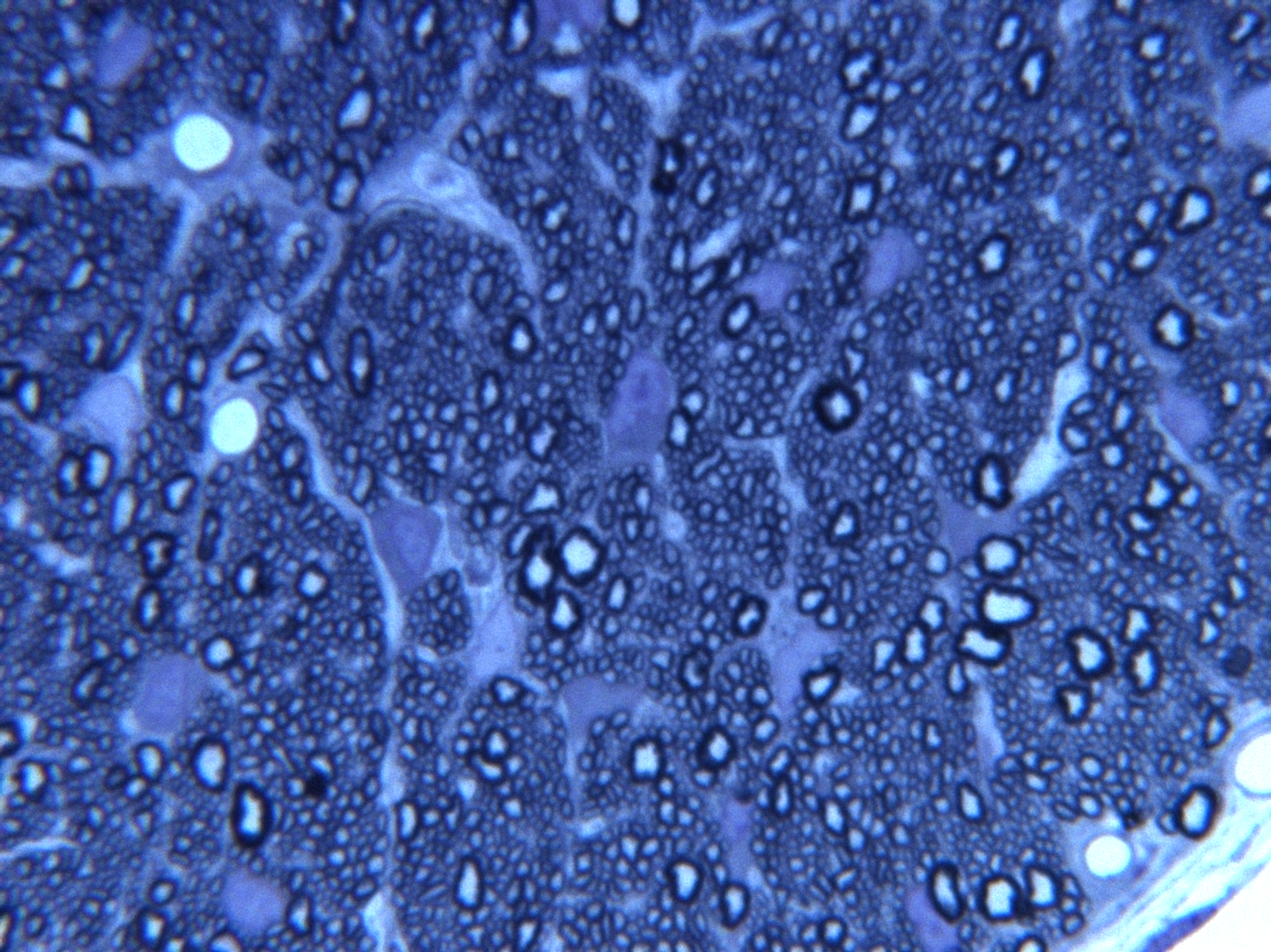
Lo primero que hice fue convertirlo a escala de grises desde la línea de comandos utilizando imagemagick
convert [[images/n_opt.png]] -colorspace gray [[images/n_opt_grayscale.png]]

Ahora abrimos ImageJ (versión 1.51j8). (Hay que instalar los plugins https://imagescience.org/meijering/software/featurej/ y http://example.com y reiniciarlo la primera vez)
Abrimos la imagen en escala de grises.
Ahora corremos el algoritmo CLAHE dos veces. Está en Plugins-CLAHE. Usamos los defaults (blocksize=127, histogram bins=256, max slope=3.0). La imagen queda mucho más homogénea.

Tenemos que encontrar un umbral que separe membranas/mielina del resto de la imagen. Para esto, vamos a Image-Adjust-Auto local threshold. Seleccionamos el método Bernsen con radio=15 y dejamos seleccionado White objects on black bacground. Nota: He probado solo parcialmente los otros métodos, y éste ha dado buen resultado, aunque no descarto que otro pueda ser mejor.
La imagen quedará binarizada.

Ahora sí ya solo queda encontrar los axones. Usaremos Analyze-Analyze particles. En Circularity seleccionamos 0.5-1, lo que hace al algoritmo buscar partículas semi o casi ciruclares, e ignorar cosas lineales. Aquí debemos tener cuidado cuando tengamos un corte no perfectamente perpendicular a los axones. Además, debemos jugar un poco con el tamaño de la partícula, que aquí está de cero a infinito. Quizás con ésto podemos controlar más el algoritmo, y/o dividir los axones chicos de los grandes. Esto y las estadísticas están mejor si tenemos claro las dimensiones de cada pixel en micras y se lo decimos a ImageJ dimensions.

El resultado son los perfiles del lúmen de los axones, y una tabla donde se resumen sus áreas.

NOTA Para especificar las dimensiones físicas de una imagen cargada en ImageJ:
Cargar la imagen.
Image-Properties.
Unit of length = um Pixel width&height=0.0645 (ejemplo, no es siempre así, útil para probar magnificación 100X).
Tabla de contenidos
- Home
- Como colaborar en la Wiki
- rocket.chat
- Resonadores
- Bash
- Clúster
- Procesamiento de Imágenes
- fMRI
- DW-MRI
- FIJI - Análisis histológico
- Herramientas Software
- Otros